谢承志 李春香 崔银秋 张全超 周慧 朱泓 | 尼雅遗址古代居民线粒体DNA研究
尼雅遗址是《汉书·西域传》中记载的“精绝”国故址,是丝绸之路沿线保存最完好的古代遗址之一,14C测定在距今1480~2635年之间。本实验对一例尼雅股骨样本线粒体高可变一区(364bp)进行扩增和测序,并同时做了编码区的限制性片段长度分析,结果显示这一个体属于U3亚型。U3亚型在现代人群中主要集中分布在近东和伊朗,与其他新疆古代人群对比显示:尼雅人群和山普拉人群可能存在一定的母系遗传联系,与潘其凤先生的体质人类学分析结果一致。
古DNA研究是以分子生物学技术为基础发展起来的一个新兴领域,对其研究能够打破时间的限制,直接分析古代材料中的遗传信息,通过比较现代生物和古生物之间的差异和联系,研究包括人类在内的各种生物的起源、进化和迁徙等问题。[2]最近古DNA研究取得了一些重要进展,Haak等发表了对德国、奥地利和西班牙地区出土的57例距今7500年前的欧洲农民的古DNA分析结果,表明现代欧洲人的祖先不可能是新石器时期的农民,这为现代欧洲人起源于旧石器时期的猎户采集人提供了佐证。[3]随后Nature和Science又先后发表了猛犸象化石中得到的DNA数据,Krause等通过猛犸象的线粒体DNA全基因组数据证明猛犸象与亚洲象有最近的亲缘关系,这些研究给未来古DNA的发展指明了方向。[4]
尼雅遗址位于新疆民丰县城以北100多公里的塔克拉玛干沙漠南缘,遗址以北纬37°58′34″19、东经82°43′14″92的佛塔为中心,面积约170 余平方公里。[5]尼雅遗址为汉晋时期精绝国的故址,精绝国是古代沿丝绸之路的西域南道形成的聚落之一,作为东西交通的要地曾繁荣一时,在东汉时曾一度被于阗所吞并。[6]尼雅遗址在1901年被英国探险家斯坦因首先发现,他在尼雅发现的大量珍贵文物轰动了整个欧洲学术界,称尼雅为东方“庞培城”。1995年10月,中日两国考古学家深入塔克拉玛干沙漠,开始了对尼雅遗址的大规模科学考察。此次发掘是近一个世纪以来收获最为丰硕的一次,被评为“95全国十大考古新发现”之一。这次考古价值最高的发现是大量保存完好、特色鲜明的织锦和写有佉卢文的木简函牍,出土文物之丰富、保存之完好震惊了中国乃至世界考古界。在尼雅出土的文物中,人们同时看到了西亚风格的玻璃器、希腊风格的艺术品、犍陀罗的装饰图案、印度的棉织物、黄河流域的锦绢漆器、铜镜、纸片等。难怪许多学者说,尼雅是古中国、古印度、古希腊和古波斯四大古代文明的一个罕见交汇点。
我们选择了一例尼雅遗址出土的样本进行线粒体DNA高可变一区(Hypervariable Region I,HVR-I)的测序,以及编码区的限制性片段长度多态性分析(restriction fragment length polymorphism,RFLP)。由此来分析该尼雅个体与现代欧亚大陆居民以及古代新疆居民间的母系遗传关系。
1.样本与处理
我们选取了一例尼雅遗址出土的保存完好的股骨,体质人类学家鉴定属于一35~45岁男性个体。将样本在紫外线下照射45 min,然后打磨去表面2~3 mm的薄层以去除表面污染,液氮保存过夜后用球磨机(FRITSCH,German)打磨成骨粉,骨粉保存在4℃。[7]
2.DNA的提取、扩增及测序
DNA的提取使用GENECLEANⓇfor Ancient DNA试剂盒(BIO101,USA),提取液用Uracil-N-Glycosylase(UNG)在37℃处理30 min,以去除由胞嘧啶水解脱氨变成尿嘧啶所产生的损伤突变。[8]
使用四对套叠引物扩增线粒体DNA高可变一区16035~16398长度共364bp的序列。为了做出准确的线粒体单倍型分析,还选取了3对引物扩增线粒体DNA的编码区进行限制性片段长度多态性分析(表1)。扩增产物使用2%琼脂糖(Biowest,German)进行检测,所得DNA片段用QIAEXⓇⅡGEL Extraction Kit(QIAGen,German)纯化,最后使用ABI PRISMTM310 Genetic Analyzer(Applied Biosystems,USA)测序。
表1 DNA扩增中使用的引物

3.防污染措施
实验中严格按照Cooper(2000)和Poina(2004)等人提出的古DNA操作规范进行。[9]古DNA研究在一个专门的古DNA实验室中进行,实验区域完全独立于其他现代分子生物学实验区域,并有正压的空气过滤系统,紫外线(254nm)照射,实验器材都经过高温蒸汽灭菌,并用DNA—OffMTM(Q·BIO gene,USA)处理不能灭菌的仪器表面。每次PCR都要有抽提空白和PCR空白,来判断是否有外源污染。对尼雅样本进行了三次独立抽提,由三次抽提产物的扩增序列来验证结果真实性。所得序列与实验室所有人员的线粒体DNA高可变一区的序列进行比对,以排除实验室人员污染的可能。
从尼雅样本中得到了真实可靠的线粒体高可变一区364bp的序列(16035~16398),三次抽提产物的扩增序列结果一致,未发现来自实验室人员的污染。该序列与剑桥标准序列(Cambridge Reference Sequence,CRS)[10]相比,共检测出两个变异位点,分别是16260 C―T和16343 A―G的转换。RFLP分析结果如下:-10397 Alu I, +12703 Mbo II 和+12308 Hinf I。从RFLP分析结果及高可变一区序列特征分析,尼雅样本应属于单倍型类群U下的U3亚型。[11]
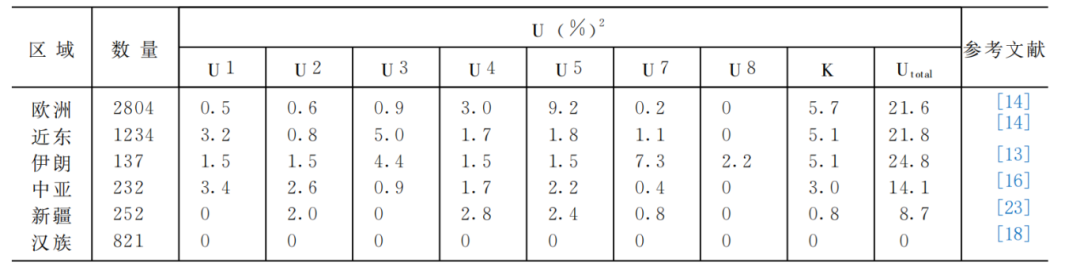
线粒体的单倍型类群U由十个亚型构成,包括“U1”—“U9”型和K型。[12]根据Quintana-Murci等人提出的分型原则,[13]我们计算了U型及其亚型在对比人群中的分布情况(表2)。从表中可以看出,U型分布最高的是在欧洲(21.6%)、近东(21.8%)[14]和伊(24.8%)[15],在中亚(14.1%)[16]和新疆(8.7%)[17]的分布逐渐减少,在汉族人群中还未被发现。[18]U型在欧亚大陆的分布呈现明显的由西向东的递减过程。
表2 U型及其亚型在对比人群中的分布情况
*单倍型类群基于Quintana-Murciet al.(2004)[19]划分
U3亚型的分布比单倍型类群U的分布更局限,U3亚型主要分布在近东(5.0%)[20]和西亚(4.4%)[21],在中亚和欧洲的分布很少,都是在0.9%左右,[22]在现代新疆和汉族,这一亚型从未被发现过。[23]由此可见,除近东和伊朗外,U3亚型在欧亚大陆其他区域都很少分布。现代新疆人群的线粒体DNA数据很少,而且主要集中在喀什和伊犁,虽然在目前的现代新疆数据中还没发现过U3亚型,[24]但受到样本数量和地域的限制,并不能完全排除U3亚型在现代新疆分布的可能。潘其凤先生的体质人类学研究结果表明,尼雅遗址古代居民的体质特征大部分与印度-地中海人种支系中的印度-阿富汗类型(即欧罗巴人种地中海东支)接近。[25]该尼雅样本确定的U3亚型主要集中在近东和伊朗人群中,这两个地区的居民在人类学分析上也主要属于欧罗巴人种地中海东支,可见,潘其凤先生的研究结果也是对本实验结果的有力佐证。
为了从分子生物学的角度来分析尼雅人群与其他新疆古代人群的遗传关系,我们分析了U3亚型在其他古代人群中的分布情况。结果只在山普拉人群(2200~1800 YBP)中发现了4例个体属于U3亚型(未发表数据),其他人群都没有发现这一亚型,例如:察吾呼(2500~3000 YBP)[26]、 吐鲁番(2300~2600 YBP)[27]和罗布诺尔(3800~4000 YBP)。[28]这一在现代新疆和古代新疆都很少分布的类型在尼雅和山普拉人群中都有发现,可见这两个人群可能存在一定的母系遗传关系。潘其凤先生通过对比尼雅人群与新疆其他地区古代人群的人类学资料显示尼雅遗址古代居民的体质特征与山普拉组最为接近,[29]与本实验通过分子生物学方法得到的结果一致。
尼雅人群和山普拉人群都发现了U3亚型,可能有以下三种推测:第一,这两个人群可能有共同的母系祖先,是从同一个祖先人群繁衍来的;第二,尼雅属于古代精绝国遗址,山普拉属于古代于阗国遗址,在东汉时期精绝国曾一度被于阗国所吞并,也许就是这一时期人群间的基因交流结果;最后,山普拉和尼雅都位于丝绸之路南线上,而且这两个遗址的年代也都处在丝绸之路最鼎盛的时期,这一单倍型类群也许是由来自近东或伊朗并定居在这里的商人遗传下来的。当然,最后一种推测的可能性并不大,因为古代往来丝绸之路的商人以男性为主,由母系遗传的线粒体DNA并不会受到来自男性的影响,同时山普拉人群中的U3亚型的分布频率非常高(13例样本中发现了4例U3亚型),即使有来自近东或伊朗的女性也不会对当地人群的线粒体DNA单倍型类群分布产生这么大的影响。
当然,由于样本数量的限制,对于尼雅人群的起源问题还不能做出确切的推断。但通过U3亚型在现代人群及新疆古代人群的对比,我们至少可以得出以下结论:1.现代欧亚大陆人群的U3亚型分布频率的对比显示,这一亚型主要分布在地中海以东的近东和伊朗人群中;2.与古代新疆人群的对比显示,只有山普拉人群发现了4例U3个体,这可能暗示尼雅人群和山普拉人群存在一定的母系遗传联系。以上两点分析与潘其凤先生的研究结果一致,为体质人类学分析提供了来自分子生物学的证据。
[1]本文的研究得到了国家基础科学人才培养基金资助(项目批准号:J0530184)。
[2] PääboS,Poinar HN,Serre D,Genetic Analyses from Ancient DNA Annu Rev Genet,2004,38:pp.645—679.Hofreiter M,Serre D,Poinar HN,Ancient DNA Nat Rev Genet,2001,2:pp.353—359.
[3] Haak W,Forster P,Bramanti B,Ancient DNA from the First European Farmers in 7500—year—oldNeolithic Sites Science,2005,310:pp.1016—1018.
[4] Krause J,Dear PH,Pollack JL. Multiplex Amplification of the mammoth Mitochondrial Genome and the Evolution of Elephantidae Nature,2005,439:pp.724—727. Poinar HN,Schwarz C,Qi J,Metagenomics to Paleogenomics:Large—scale Sequencing of Mammoth DNA Science,2005,311:pp.392—394.
[5]中日·日中共同尼雅遗迹学术考察队:《中日·日中共同尼雅遗迹学术调查报告第二卷》,日本:中村印刷株式会社,1999年,第3~4页。
[6]《中日·日中共同尼雅遗迹学术调查报告第二卷》,第304~311页。
[7]谢承志、刘树柏、崔银秋等:《新疆察吾呼古代居民线粒体DNA研究》,载《吉林大学学报(理学版)》2005年第4期。
[8] Homas M,Gilbert P,Willerslev E,Distribution Patterns of Postmortem Damage in Human Mitochondrial DNA Am J Hum Genet,2003,72:pp.32—47.
[9] Cooper A. Poinar HN.Ancient DNA:Do It Right or Not At all Science,2000,289:pp.1139.Poinar HN.Criteria for Authenticity in Ancient DNA Work New York:Taylor and Francis Group,2004,pp.115—116.
[10] Anderson S,Bankier AT,Barrell BG,Sequence and Organization of the Human Mitochondrial Genome Nature,1981,290:pp.457—465.
[11] Quintana—Murci L,Chaix R,Wells RS,Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[12] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.Achilli A,Rengo C,Battaglia V,Saami and Berbers—An Unexpected Mitochondrial DNA Link Am J Hum Genet,2005,76:pp.883—886.
[13] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[14] Richards M,Macaulay V,Hickey E,Tracing European Founder Lineages in the Near Eastern mtDNA Pool Am J Hum Genet,2000,67:pp.1251—1276.
[15] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[16] Comas D,Plaza S,Wells RS,Admixture,Migrations,and Dispersals in Central Asia:Evidence from Maternal DNA Lineages Eur J Hum Genet,2004,12:pp.495—504.
[17] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[18] Yao YG,Kong QP,Bandelt HJ,Phylogeographic Differentiation of Mitochondrial DNA in Han Chinese Am J Hum Genet,2002,70:pp.635—651.
[19] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[20] Tracing European Founder Lineages in the Near Eastern mtDNA Pool Am J Hum Genet,2000,67:pp.1251—1276.
[21] Where West Meets East:The Complex mt DNA Landscape of the Southwest and Central Asian Corridor Am J Hum Genet,2004,74:pp.827—845.
[22] Tracing European Founder Lineages in the Near Eastern mtDNA Pool Am J Hum Genet,2000,67:pp.1251—1276.Admixture,Migrations, and Dispersals in Central Asia:Evidence from Maternal DNA Lineages Eur J Hum Genet,2004,12:pp.495—504.
[23] Yao YG,Kong QP,Wang CY,Different Matrilineal Contributions to Genetic Structure of Ethnic Groups in the Silk Road Region in China Mol Biol Evol,2004,21:pp.2265—2280. Phylogeographic Differentiation of Mitochondrial DNA in Han Chinese Am J Hum Genet,2002,70:pp.635—651.
[24]Different Matrilineal Contributions to Genetic Structure of Ethnic Groups in the Silk Road Region in China Mol Biol Evol,2004,21:pp.2265—2280.
[25]《中日·日中共同尼雅遗迹学术调查报告第二卷》,第161~172页。
[26]《新疆察吾呼古代居民线粒体DNA研究》,载《吉林大学学报(理学版)》2005年第4期。
[27]崔银秋、段然慧、周慧等:《新疆古代居民的遗传结构分析》,载《高等学校化学学报》2002年第12期。
[28]崔银秋、许月、杨亦代等:《新疆罗布诺尔地区铜器时代古代居民mtDNA多态性分析》,载《吉林大学学报(医学版)》2004年第4期。
[29]《中日·日中共同尼雅遗迹学术调查报告第二卷》,第161~172页。
